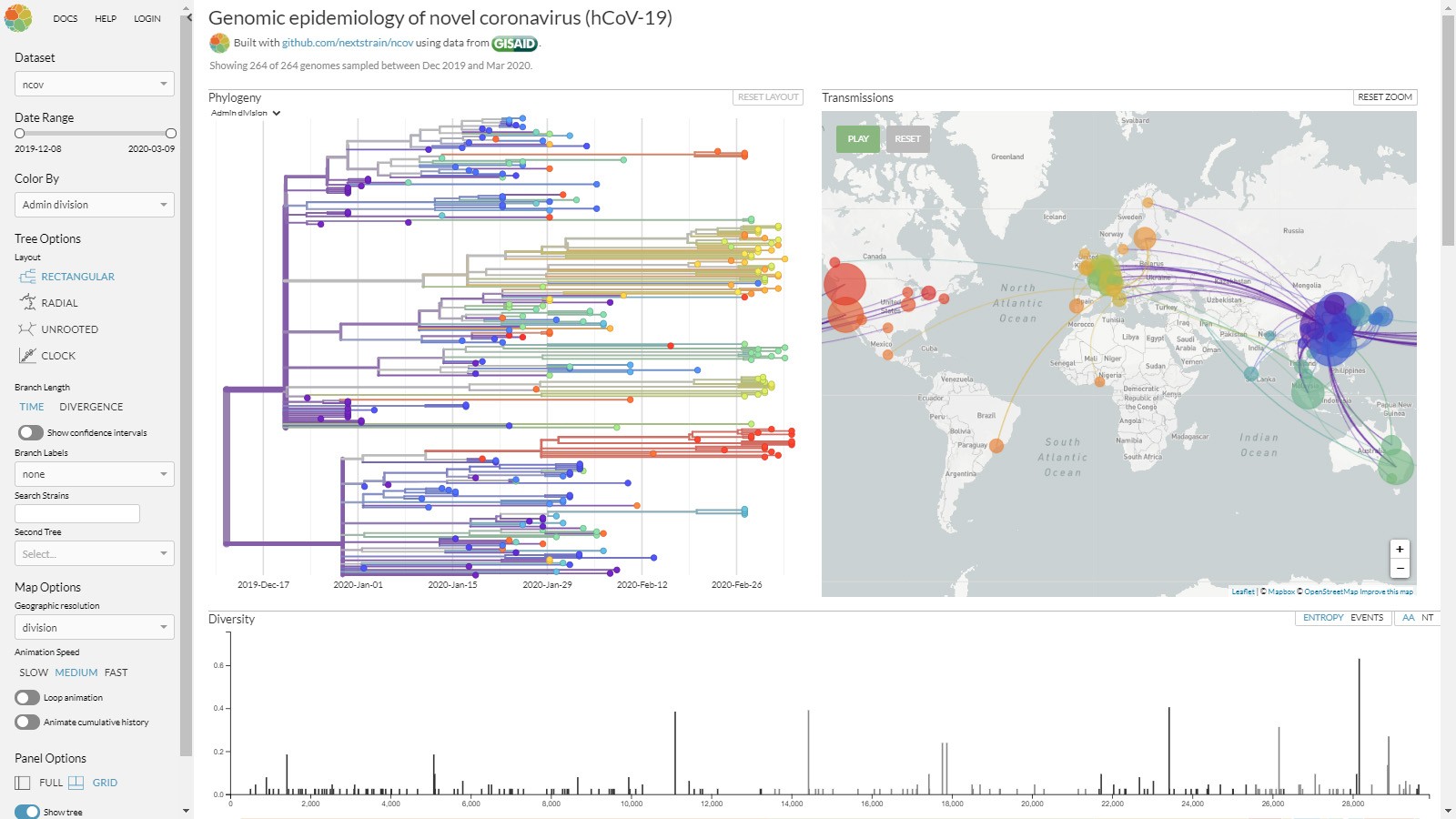
Restaurants, bars : le protocole sanitaire confirmé par le gouvernement s’appliquera dès le 19 mai.
Afin de reprendre leurs activités, les commerces vont de nouveau pouvoir accueillir du public mais sous certaines conditions sanitaires. Un protocole sanitaire a été mis en place pour organiser la réouverture le 19 mai 2021.